- 1. 使用更快的内核
- 2. 更高效的使用pandas
- 2.1. 高效数据IO
- 2.2. 高效遍历数据--避免常规循环
- 2.3. 高效数据拼接
- 2.4. 高效索引
- 2.5. 能改成numpy 就numpy
- 2.6. 多线程并行计算
- 3. 更小的pandas
- 4. 使用numpy 代替
- 5. 参考资料
1. 使用更快的内核
1.1. 使用Cython
在jupyter notebook 中使用 Cython magic function2 可以加速X3倍,对高度使用pandas的代码优化效果不明显。
原理
对python 代码转C进行提前编译,对for 循环等python解释器执行的代码进行优化,对高度使用pandas的代码优化效果不明显。
# Cell 1 %load_ext Cython # Cell 2 ## 注意需要添加相应的依赖项/包 %%cython import package def fun_name(foo,boo): ... return res # Cell 3 df.apply(lambda x:fun_name(x))
1.2. 使用 numba
numba.njit:更高的效率
当循环被认为可行时,通常会通过numba底层的NumPy数组对其进行优化,以尽可能多地移至C。
实际上,将numba性能提高到了微秒。没有一些繁琐的工作,将很难获得比这更高的效率。
from numba import njit @njit def divide(a, b): res = np.empty(a.shape) for i in range(len(a)): if b[i] != 0: res[i] = a[i] / b[i] else: res[i] = 0 return res %timeit divide(df['A'].values, df['B'].values) # 717 µs
使用@njit(parallel=True)可以为更大的阵列提供进一步的提升
1.3. 更快的numpy内核--BLAS库加速
BLAS(basic linear algebra subroutine) 是一系列基本线性代数运算函数的接口(interface)标准.
常见的BLAS库
BLAS 的官网是 Netlib, 可以浏览完整的说明文档以及下载源代码. 这个版本的 BLAS 被称为 reference BLAS, 运行速度较慢, 通常被其他版本用于衡量性能.
英特尔数学核心库(Intel MKL) 的开发是致力于加速在intel处理器上进行的数学运算。该库支持 Windows, Linux and macOS 操作系统。
查看
安装
pip install intel-numpy conda install numpy # conda 版本已经是优化过的 Anoconda (base)环境里面已经安装过
查看
import numpy as np np.show_config() >>> # No MKL blas_mkl_info: NOT AVAILABLE blis_info: NOT AVAILABLE openblas_info: library_dirs = ['C:\\projects\\numpy-wheels\\numpy\\build\\openblas'] libraries = ['openblas'] language = f77 define_macros = [('HAVE_CBLAS', None)] blas_opt_info: library_dirs = ['C:\\projects\\numpy-wheels\\numpy\\build\\openblas'] libraries = ['openblas'] language = f77 define_macros = [('HAVE_CBLAS', None)] lapack_mkl_info: NOT AVAILABLE openblas_lapack_info: library_dirs = ['C:\\projects\\numpy-wheels\\numpy\\build\\openblas'] libraries = ['openblas'] language = f77 define_macros = [('HAVE_CBLAS', None)] lapack_opt_info: library_dirs = ['C:\\projects\\numpy-wheels\\numpy\\build\\openblas'] libraries = ['openblas'] language = f77 define_macros = [('HAVE_CBLAS', None)] # with mkl mkl_info: libraries = ['mkl_rt'] library_dirs = ['D:/ProgramData/Anaconda3\\Library\\lib'] define_macros = [('SCIPY_MKL_H', None), ('HAVE_CBLAS', None)] include_dirs = ['C:\\Program Files (x86)\\IntelSWTools\\compilers_and_libraries_2019.0.117\\windows\\mkl', 'C:\\Program Files (x86)\\IntelSWTools\\compilers_and_libraries_2019.0.117\\windows\\mkl\\include', 'C:\\Program Files (x86)\\IntelSWTools\\compilers_and_libraries_2019.0.117\\windows\\mkl\\lib', 'D:/ProgramData/Anaconda3\\Library\\include'] blas_mkl_info: libraries = ['mkl_rt'] library_dirs = ['D:/ProgramData/Anaconda3\\Library\\lib'] define_macros = [('SCIPY_MKL_H', None), ('HAVE_CBLAS', None)] include_dirs = ['C:\\Program Files (x86)\\IntelSWTools\\compilers_and_libraries_2019.0.117\\windows\\mkl', 'C:\\Program Files (x86)\\IntelSWTools\\compilers_and_libraries_2019.0.117\\windows\\mkl\\include', 'C:\\Program Files (x86)\\IntelSWTools\\compilers_and_libraries_2019.0.117\\windows\\mkl\\lib', 'D:/ProgramData/Anaconda3\\Library\\include'] blas_opt_info: libraries = ['mkl_rt'] library_dirs = ['D:/ProgramData/Anaconda3\\Library\\lib'] define_macros = [('SCIPY_MKL_H', None), ('HAVE_CBLAS', None)] include_dirs = ['C:\\Program Files (x86)\\IntelSWTools\\compilers_and_libraries_2019.0.117\\windows\\mkl', 'C:\\Program Files (x86)\\IntelSWTools\\compilers_and_libraries_2019.0.117\\windows\\mkl\\include', 'C:\\Program Files (x86)\\IntelSWTools\\compilers_and_libraries_2019.0.117\\windows\\mkl\\lib', 'D:/ProgramData/Anaconda3\\Library\\include'] libraries = ['mkl_rt'] library_dirs = ['D:/ProgramData/Anaconda3\\Library\\lib'] define_macros = [('SCIPY_MKL_H', None), ('HAVE_CBLAS', None)] include_dirs = ['C:\\Program Files (x86)\\IntelSWTools\\compilers_and_libraries_2019.0.117\\windows\\mkl', 'C:\\Program Files (x86)\\IntelSWTools\\compilers_and_libraries_2019.0.117\\windows\\mkl\\include', 'C:\\Program Files (x86)\\IntelSWTools\\compilers_and_libraries_2019.0.117\\windows\\mkl\\lib', 'D:/ProgramData/Anaconda3\\Library\\include'] lapack_opt_info: libraries = ['mkl_rt'] library_dirs = ['D:/ProgramData/Anaconda3\\Library\\lib'] define_macros = [('SCIPY_MKL_H', None), ('HAVE_CBLAS', None)] include_dirs = ['C:\\Program Files (x86)\\IntelSWTools\\compilers_and_libraries_2019.0.117\\windows\\mkl', 'C:\\Program Files (x86)\\IntelSWTools\\compilers_and_libraries_2019.0.117\\windows\\mkl\\include', 'C:\\Program Files (x86)\\IntelSWTools\\compilers_and_libraries_2019.0.117\\windows\\mkl\\lib', 'D:/ProgramData/Anaconda3\\Library\\include']
比较
比较OpenBLAS,Intel MKL和Eigen的矩阵相乘性能
EIGEN: 是一个线性算术的C++模板库。功能强大、快速、优雅以及支持多平台,可以使用该库来方便处理一些矩阵的操作,达到类似matlab那样的快捷。 需要定义 EIGEN_NO_DEBUG 阻止运行时assertion。编译单线程版本需要开启 -DEIGEN_DONT_PARALLELIZE. 在试验中,我们采用 EIGEN 原生 BLAS 实现。
Intel MKL: 英特尔数学核心函数库是一套经过高度优化和广泛线程化的数学例程,专为需要极致性能的科学、工程及金融等领域的应用而设计。它可以为当前及下一代英特尔处理器提供性能优化,包括更出色地与 Microsoft Visual Studio、Eclipse和XCode相集成。英特尔 MKL 支持完全集成英特尔兼容性 OpenMP 运行时库,以实现更出色的 Windows/Linux 跨平台兼容性。在试验中的多线程版本需要链接到 mkl_gnu_thread,而不是 mkl_intel_thread,单线程版本需要链接到 mkl_sequential_thread。
OpenBLAS: 是一个高性能多核 BLAS 库,是 GotoBLAS2 1.13 BSD 版本的衍生版。OpenBLAS 的编译依赖系统环境,并且没有原生单线程版本,在实验这哦那个,通过设置 OMP_NUM_THREADS=1 来模拟单线程版本,可能会带来一点点的性能下降。
每个测试程序的编译都采用 “-O4 -msse2 -msse3 -msse4” 优化, 通过设置 OMP_NUM_THREADS 来控制使用的线程数量. 除了 OpenBLAS,其他两个库的测试程序都分别有单线程和多线程的编译版本。
如果MKL编译出现问题,建议参考Intel Math Kernel Library Link Line Advisor
单线程版本
我在实验中进行了一系列的非稀疏矩阵相乘运算,矩阵规模也逐渐增大,单线程的运行时间如下表所示,其中采用的测试轮数为5轮,其中红色表示性能最好的一组实验结果。
Matrix-Dimension Eigen MKL OpenBLAS
500 0.04159 0.03122 0.03058
1000 0.31789 0.24339 0.23730
1500 1.04589 0.81445 0.79869
2000 2.37567 1.92036 1.87102
2500 4.68266 3.78569 3.64548
3000 8.28073 6.42630 6.29797
3500 13.07470 10.25096 9.98417
4000 19.34550 15.21931 14.87500
4500 27.52767 21.45024 21.18227
5000 37.67552 29.31631 29.07229
从图中可以看出,OpenBLAS的性能最好,MKL的表现也很不错,而EIGEN的表现却很糟糕。
多线程版本
在多线程的测试中,我们采用多个CPU核心来做矩阵乘法运算,所有的结果也同样采用5轮训练,我们采用的CPU核数分别是8,16,32,48。
Cores = 8
Matrix-Dimension Eigen MKL OpenBLAS
1000 0.05658 0.03955 0.06468
2000 0.34981 0.26200 0.23879
3000 1.20781 0.85449 0.80737
4000 2.65490 1.90273 1.88366
5000 5.03304 3.73005 3.67966
6000 8.78654 6.52766 6.31980
7000 13.55611 10.13758 10.07120
8000 19.81634 15.03530 14.89440
9000 29.11329 21.54359 21.26992
10000 39.01563 29.93075 29.22034
Cores = 16
Matrix-Dimension Eigen MKL OpenBLAS
1000 0.05708 0.02185 0.03897
2000 0.26694 0.13807 0.30461
3000 0.70686 0.43692 0.93511
4000 1.45129 0.97720 2.06761
5000 2.59477 1.90665 2.49280
6000 5.43438 3.30945 7.01299
7000 8.01124 5.17896 6.84496
8000 11.22280 7.81439 12.99240
9000 15.15625 11.08906 21.82488
10000 19.91151 15.22039 30.86908
Cores = 32
Matrix-Dimension Eigen MKL OpenBLAS
1000 0.04003 0.02792 0.02244
2000 0.51213 0.14363 0.16990
3000 1.13647 0.51105 0.54635
4000 1.58793 1.10219 1.26401
5000 2.88341 2.07923 2.48735
6000 5.92779 3.42785 4.26794
7000 7.91650 5.32176 6.69391
8000 11.96467 7.65395 9.98951
9000 17.45420 10.28328 14.14108
10000 23.31314 15.10077 19.34171
Cores = 40
Matrix-Dimension Eigen MKL OpenBLAS
1000 0.03691 0.02877 0.01779
2000 0.37739 0.14037 0.13655
3000 0.61183 0.41057 0.44113
4000 2.43670 1.02625 1.01414
5000 3.18099 1.91092 1.97898
6000 8.24002 2.96157 3.40685
7000 11.59889 4.68312 5.38634
8000 9.50613 6.98434 7.95971
9000 14.83066 9.60891 11.37585
10000 23.67187 15.52151 15.52680
Cores = 48
Matrix-Dimension Eigen MKL OpenBLAS
1000 0.03635 0.02398 0.01548
2000 0.36417 0.13408 0.11496
3000 2.32388 0.39291 0.36669
4000 2.32030 1.13244 0.85790
5000 2.08269 1.75812 1.66785
6000 8.70766 2.98694 2.85609
7000 8.23543 4.62340 4.53257
8000 21.18603 6.68886 6.72820
9000 19.86504 9.59635 9.50597
10000 16.10920 13.13038 13.04432
可以看出,MKL和OpenBLAS都提供了比较好的性能,MKL性能还更好一点,在各别多线程条件下了,可能某些原因或者我机器设置的问题,出现了各别性能异常,比如小矩阵运算时间反倒比大矩阵运算长,或者更多的线程却不能提供更好的性能。这些情况后面可能还需要查一查。
伸缩性
另外,我也测试了使用不同的cpu核数对性能的影响,下面两个图描述了把cpu从1增加到20的条件下,5000×5000的矩阵相乘的时间开销和加速比。
结论
就我的测试环境而言,Intel MKL 和 OpenBLAS 似乎是矩阵相乘运算方面性能最佳的 BLAS 库,在多核以及不同规模的矩阵方面都具有较好的伸展性和稳定性,而对于单线程情况,OpenBLAS相比 MKL 在性能上有一定提升。
2. 更高效的使用pandas
2.1. 高效数据IO
2.1.1. 使用pandas自带函数读取文件
pandas是带buffer,按块读取的。python直接读取是按字节的。一般情况下使用pandas会比直接使用python快
2.1.2. 使用二进制格式文件进行I/O
选择合适的文件格式,有利于IO性能提升5
在极度追求IO性能的情况下推荐使用二进制文件格式(如:hdf、feather或h5py格式)。
# Text csv df=pd.read_csv("./test.csv") # 41.1 s df.to_csv("./test.csv",index=True) # 4min 7s # 800MB -> ./test.csv # 1.6GB -> df
2.1.2.1. HDF5 Format
HDF5 是一种高效的文件格式,
I/O 读取需要依赖 PyTables >= 3.0.0.
# binary HDF5 Format df=pd.read_hdf("./test.h5",key="df") # 6.39 s df.to_hdf("./test.h5",key="df",model="w") # 48.3 s # 1.6GB -> ./test.h5 # 1.6GB -> df
2.1.2.2. feather
Feather 的开发依赖于 Apache Arrow 计划,是一种快速、互动的文件存储格式
注意:
- pd.read_feather 默认 nthreads=1,可以调整
# feather df=pd.read_feather("./test.feather",nthreads =1) # 21.9 s df=pd.read_feather("./test.feather",nthreads =4) # 17s df.to_feather("./test.feather") # 8 s # 1.6GB -> ./test.feather # 1.6GB -> df
2.1.2.3. parquet--推荐
apache parquet 为dataframe 格式的内存数据,IO处理提供了优秀的解决方案。
pd.read_parquet 支持多引擎,兼顾持久化后的内存,支持多种文件路径
注意:
1. columns_name 不支持重复的列名和非字符串列名。
2. df.index.name=None or "" index 指引如果有,必须是字符串
关于引擎的说明:
1. fastparquet uses numba,
2. pyarrow uses a c-library,不支持 datetime数据
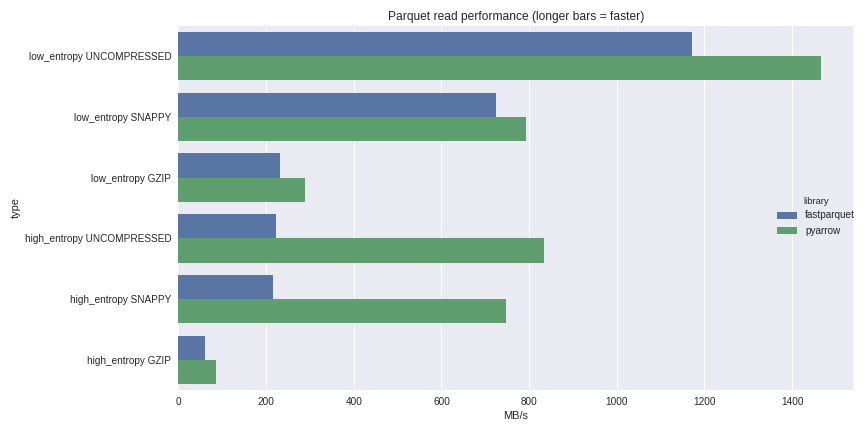
df=pd.read_parquet('./test.parquet', engine='pyarrow') # 13.9 s df.to_parquet('./test.parquet', engine='pyarrow') # 12 s # 7.13 MB -> ./test.parquet # 1.6GB -> df # 推荐使用 pyarrow 引擎 df=pd.read_parquet('./test.parquet', engine='fastparquet') df.to_parquet('./test.parquet', engine='fastparquet',compression="UNCOMPRESSED") # 8.26 s # 1.6GB-> ./test.parquet df.to_parquet('./test.parquet', engine='fastparquet',compression="GZIP") # 7.13 MB -> ./test.parquet # 14.7s # 1.6GB -> df
2.1.2.4. Python Pickle Format
直接使用
# binary Python Pickle Format df=pd.read_pickle("./test.pkl") # 3.46 s df.to_pickle("./test.pkl") # 35.7 s # 1.6GB -> ./test.pkl # 1.6GB -> df
一般情况下HDF的读取比读取csv文件快几十倍,但HDF文件在大小上会稍微大一些。
比较
print("pandas df to disk ####################################################") print('example_feather:') %timeit feather.write_feather(df, 'example_feather') # 2.62 ms ± 35.8 µs per loop (mean ± std. dev. of 7 runs, 100 loops each) print('example_parquet:') %timeit pq.write_table(pa.Table.from_pandas(df), 'example.parquet') # 3.19 ms ± 51 µs per loop (mean ± std. dev. of 7 runs, 100 loops each) print() print("for comparison:") print('example_pickle:') %timeit df.to_pickle('example_pickle') # 2.75 ms ± 18.8 µs per loop (mean ± std. dev. of 7 runs, 100 loops each) print('example_fp_parquet:') %timeit fp.write('example_fp_parquet', df) # 7.06 ms ± 205 µs per loop (mean ± std. dev. of 7 runs, 1 loop each) print('example_hdf:') %timeit df.to_hdf('example_hdf', 'key_to_store', mode='w', table=True) # 24.6 ms ± 4.45 ms per loop (mean ± std. dev. of 7 runs, 100 loops each) print() print("pandas df from disk ##################################################") print('example_feather:') %timeit feather.read_feather('example_feather') # 969 µs ± 1.8 µs per loop (mean ± std. dev. of 7 runs, 1000 loops each) print('example_parquet:') %timeit pq.read_table('example.parquet').to_pandas() # 1.9 ms ± 5.5 µs per loop (mean ± std. dev. of 7 runs, 1000 loops each) print("for comparison:") print('example_pickle:') %timeit pd.read_pickle('example_pickle') # 1.07 ms ± 6.21 µs per loop (mean ± std. dev. of 7 runs, 1000 loops each) print('example_fp_parquet:') %timeit fp.ParquetFile('example_fp_parquet').to_pandas() # 4.53 ms ± 260 µs per loop (mean ± std. dev. of 7 runs, 1 loop each) print('example_hdf:') %timeit pd.read_hdf('example_hdf') # 10 ms ± 43.4 µs per loop (mean ± std. dev. of 7 runs, 100 loops each) # pandas version: 0.22.0 # fastparquet version: 0.1.3 # numpy version: 1.13.3 # pandas version: 0.22.0 # pyarrow version: 0.8.0 # sys.version: 3.6.3 # example Dataframe taken from https://arrow.apache.org/docs/python/parquet.html
2.1.3. 数据压缩后再进行持久化
df.to_csv('output.csv.gz' , sep='|' , header=True , index=False , chunksize=100000 , compression='gzip' , encoding='utf-8') # 使用compression='gzip' 压缩文件 df.to_csv("./test.csv",index=True) # 4min 7s # 800MB -> ./test.csv # 1.6GB -> df df.to_csv("./test.csv.gz",index=True, compression='gzip',encoding='utf-8') # 5min # 9.80MB -> ./test.csv.gz # 1.6GB -> df df.to_pickle("data.pkl.compress", compression="gzip") rt = pd.read_pickle("data.pkl.compress", compression="gzip")
2.2. 高效遍历数据--避免常规循环
总结: 如果必须要使用循环,建议使用 apply; 否则的话尽量适应向量化的方法
2.2.1. 标准遍历--x1
for i in range(0,len(df)): 判定1: df["列名"]iloc[i]=value 判定2: df["列名"]iloc[i]=value
def soc_loop(df,TEAM,): df['Draws'] = 99999 for row in range(0, len(df)): if ((df['HomeTeam'].iloc[row] == TEAM) & (df['FTR'].iloc[row] == 'D')) | \ ((df['AwayTeam'].iloc[row] == TEAM) & (df['FTR'].iloc[row] == 'D')): df['Draws'].iloc[row] = 'Draw' elif ((df['HomeTeam'].iloc[row] == TEAM) & (df['FTR'].iloc[row] != 'D')) | \ ((df['AwayTeam'].iloc[row] == TEAM) & (df['FTR'].iloc[row] != 'D')): df['Draws'].iloc[row] = 'No_Draw' else: df['Draws'].iloc[row] = 'No_Game' return df df=soc_loop(df,"Arsenal")
2.2.2. 使用内置函数 iterrows()--x321倍加速
- 使用的是python level 的loop
- 实现形式是通过为新的df列添加 list来实现的,其中list通过
append遍历来实现
add_list=[] for index,row in df.iterrows(): add_list.append(func(parmaters_1,row["选择相关列名1"],row["选择相关列名2"])) df["列名"]=add_list
2.2.3. 使用内置函数 apply()--x811倍加速
apply()方法可将函数应用于dataframe特定行或列。 其本质是另一种形式的遍历loop。 但比python-level 的遍历效率更高。
函数可以由lambda方式在代码中内嵌实现,lambda函数的末尾包含axis参数,用来告知Pandas将函数运用于行(axis = 1)或者列(axis = 0),可以普通自定义函数设置。
def foo(row): print(type(row)) return sth df.apply(lambda row: foo(row["A"]), axis=1) #输入pd.Series 到函数中,效率低 df.apply(lambda row: foo(*row), axis=1, raw=True) # raw=True 输入numpy.array 到函数中,效率高 X10 # 对一行进行操作,作用于列 df.loc[选择行,:]=df.apply(lambda col: col["index_1"]+col["index_2"], axis =0) # 对一列进行操作,作用于行 df.loc[:,选择列]=df.apply(lambda row: row["选择相关列名1"]+row["选择相关列名2"], axis =1)
2.2.4. 使用groupby()--减少遍历元素
通过使用groupby()将数据元素合并分组,避免重复元素遍历,可以提高程序运行效率。
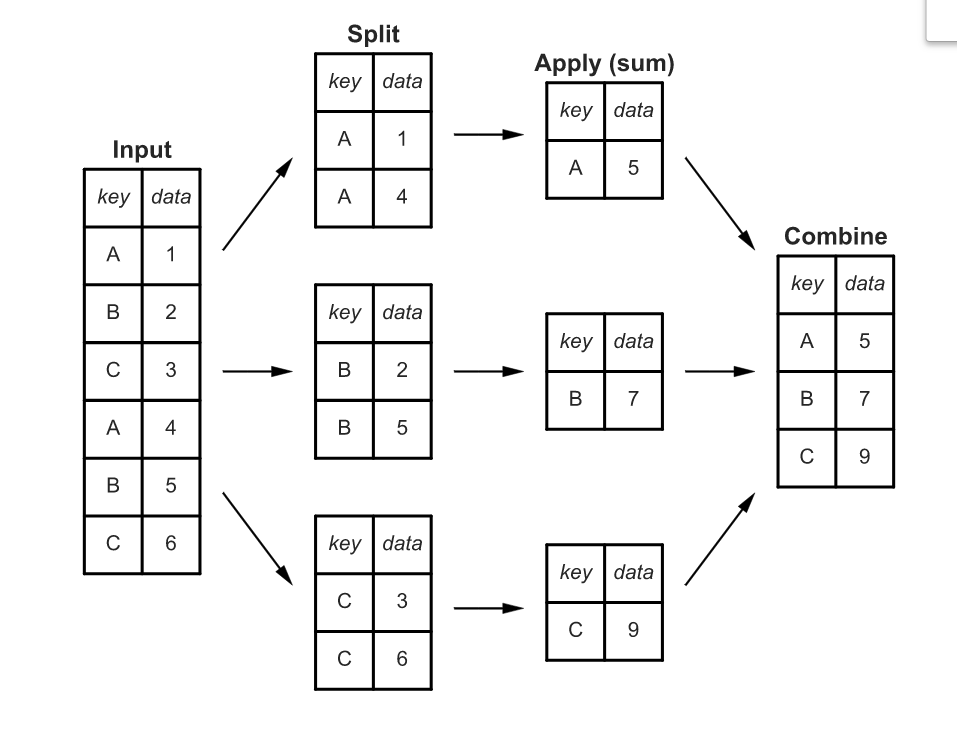
主要有:
1. grouped.apply(func)
2. grouped.agg(func)
3. grouped.transform(func)
4. grouped.uni_func() # 内置函数uni_func
上述4种应用本质上还是python-level的loop,只是针对dataframe这类的数据结构有不同程度的优化,包括底层C/C++的编译优化
2.2.4.1. groupby().apply(func)
自定义函数func
输入: apply 默认传入的是整个dataframe 到自定义函数func
原理: apply 函数将函数func 应用到您指定的每个group。
输出: apply 是一个更一般化的方法,自定义函数可以返回scalar, Series , DataFrame ,numpy array , list等形式
df = pd.DataFrame({'Q':['LI','ZHANG','ZHANG','LI','WANG'], 'A' : [1,1,1,2,2], 'B' : [1,-1,0,1,2], 'C' : [3,4,5,6,7]}) >>> Q A B C 0 LI 1 1 3 1 ZHANG 1 -1 4 2 ZHANG 1 0 5 3 LI 2 1 6 4 WANG 2 2 7 # 最好先对df 进行切片 a=df.loc[:,["A","C"]].groupby('A').apply(lambda x:x["A"]+x["C"]) # a 是 pd.Series # x 是apply 输入到自定义函数func函数中的变量,是dataframe格式 a.index=a.index.get_level_values(1) df["d"]=a >>> Q A B C d 0 LI 1 1 3 4 1 ZHANG 1 -1 4 5 2 ZHANG 1 0 5 6 3 LI 2 1 6 8 4 WANG 2 2 7 9 ###其他事项 def subtract_two(x): return x['B'] - x['C'] df.groupby('A').apply(subtract_two) # 可以正常运行 df.groupby('A').transform(subtract_two) # 报错
2.2.4.2. groupby().agg(func)
输入:agg的调用需要要指定字段,输入指定列到函数func
输出:Dataframe ,groupby index
df.groupby("Q").agg(["mean","sum"]) A B C mean sum mean sum mean sum Q LI 1.5 3 1.0 2 4.5 9 WANG 2.0 2 2.0 2 7.0 7 ZHANG 1.0 2 -0.5 -1 4.5 9 df.groupby('A').agg(dict(B='sum', C=['mean', 'prod'])) >>> B C sum mean prod A 1 0 4.0 60 2 3 6.5 42
2.2.4.3. groupby().transform(func)
自定义函数func
输入:以Series 形式输入
返回:自定义函数func 必须返回行数相同(必须与group长度相同)的一维序列(sequence,one dimensional Series, array or list,or scale),或者标量(a single scalar object )
# sequence -> same row def rand_group_len(x): return np.random.rand(len(x)) new_df=df.groupby('Q').transform(rand_group_len) new_df >>> A B C 0 0.255113 0.894918 0.518019 1 0.641294 0.849864 0.847467 2 0.815746 0.224903 0.732254 3 0.575618 0.007238 0.621828 4 0.254013 0.331561 0.431511 # scalar def group_sum(x): return x.sum() sequence=df.groupby('A').transform(group_sum) s = pd.Series(range(3)) >>> 0 0 1 1 2 2 s.transform([np.sqrt, np.exp]) >>> sqrt exp 0 0.000000 1.000000 1 1.000000 2.718282 2 1.414214 7.389056
2.2.4.4. groupby().内置函数
groupyby 可以直接用的优化过的高速运算方法有count()、sum()、prod()、mean()、median()、min()、max()、std()、var()、first()、last()。
grouop_df=df.groupby('A').mean() # grouop_df index 是A
在groupby().agg和groupby().transform时尽量使用内置函数计算,可加速计算。
grouop_df=df.groupby('A').transform("mean") # grouop_df index 是range(0,n) df.groupby('A').agg(['mean', 'std'])
2.2.5. 更快的groupby.apply()方法,使用numpy 自定义
参考4 : Fast groupby-apply operations in Python with and without Pandas
http://esantorella.com/2016/06/16/groupby/
first_category = np.random.choice(n_categories, n_obs) y = np.random.normal(0, 1, n_obs) df = pd.DataFrame({'first category': first_category, 'y': y}) class Groupby: def __init__(self, keys): """ 精华就再在这里 np.unique """ _, self.keys_as_int = np.unique(keys, return_inverse = True) self.n_keys = max(self.keys_as_int) + 1 self.set_indices() def set_indices(self): self.indices = [[] for i in range(self.n_keys)] for i, k in enumerate(self.keys_as_int): self.indices[k].append(i) self.indices = [np.array(elt) for elt in self.indices] def apply(self, function, vector, broadcast): if broadcast: result = np.zeros(len(vector)) for idx in self.indices: result[idx] = function(vector[idx]) else: result = np.zeros(self.n_keys) for k, idx in enumerate(self.indices): result[self.keys_as_int[k]] = function(vector[idx]) return result %%timeit grouped = df.groupby('first category').apply(np.mean) # 595 ms ± 162 ms a=Groupby(df["first category"].values).apply(np.mean,df["y"].values,False) # 36.7 ms ± 6.65 ms %%timeit df['mean'] = df.groupby('first category')['y'].transform(np.mean) #4.28 ms ± 1.03 ms # 还是transform好
# 充分利用这一性能对dataframe 进行分解 然后进行高效map运算 unique_keys, indices = np.unique(['a', 'b', 'a', 'a', 'b'], return_inverse = True) print(unique_keys) >>> array([{'b', 'a'}], dtype=object) print(indices) >>> #list [0, 1, 0, 0, 1] print(unique_keys[indices]) >>> array(['a', 'b', 'a', 'a', 'b'], dtype='<U1')
2.2.6. 使用向量化函数 np.vectorize代替apply 处理
np.vectorize is fake vectorize.3
np.vectorize的主要作用原理是使用python-level 的map函数封装pyfunc对输入数组求值,而不是numpy的boardcast 规则。在这里boardcast 规则并不重要。
np.vectorize 运行过程中通过使用np.frompyfunc方法将pyfunc 转换为 numpy 的通用函数ufunc,并通过一些优化(例如缓存)以实现性能提升。
np.vectorize 依然属于 Python-level 的loop,虽然它比pd.DataFrame.apply更加高效。但依据其原理,其作用效果于 map,zip 在同一级别上
%timeit list(map(divide, df['A'], df['B'])) # 43.9 ms %timeit np.vectorize(divide)(df['A'], df['B']) # 48.1 ms %timeit [divide(a, b) for a, b in zip(df['A'], df['B'])] # 49.4 ms %timeit df.apply(lambda row: divide(*row), axis=1, raw=True) # 760 ms
func=lambda x:x+2 vecfunc = np.vectorize(func) new_array = vecfunc(old_array) def foo(a,b): return a+b df['result2'] = np.vectorize(foo)(df['A'], df['B']) # func:A python function or method type(vecfunc) >>> numpy.vectorize # Vectorized function. 向量化的函数 type(new_array) >>> numpy.array
2.2.7. 使用pandas 向量化 —-x9280倍加速
pandas 向量化:
1. 避免使用python解释器 级别的loop循环
2. 使用优化的C编译器,使用内存更加高效
3. 依据有条件的切片df.loc[i,c] 及函数实现(注意实现方式)
%timeit (df['A'] / df['B']).replace([np.inf, -np.inf], 0) # 1.96 ms
# 构建函数 def func(parameter_1,col_1,col_2,..): """ col_1: df array 相同df col_2: df array 相同df """ df["列名"]=1 df.loc[行条件1,"列名"]=value_1 df.loc[行条件2(col_1==值1,col_2==值2),"列名"]=value_2 df["列名"]=func(parameter_1,df["选择列名1"],df["选择列名2"],..)
def soc_iter(TEAM,home,away,ftr): df['Draws'] = 'No_Game' df.loc[((home == TEAM) & (ftr == 'D')) | ((away == TEAM) & (ftr == 'D')), 'Draws'] = 'Draw' df.loc[((home == TEAM) & (ftr != 'D')) | ((away == TEAM) & (ftr != 'D')), 'Draws'] = 'No_Draw' df["Draws"]=soc_loop("Arsenal",df["HOME"],df["AWAY"],df["FTR"])
2.2.8. 使用numpy 向量化--x658880倍加速
pandas 向量化:
1. 避免使用python解释器 级别的loop循环
2. 使用优化的C编译器,使用内存更加高效
3. 条件切片df.loc[i,c] 及函数实现(注意实现方式)
4. 函数输入的是numpy array (不同点)
%timeit np.where(df['B'] == 0, 0, df['A'] / df['B']) # 1.17 ms # 构建函数 def func(parameter_1,col_1,col_2,..): df["列名"]=1 df.loc[行条件1,"列名"]=value_1 df.loc[行条件2(col_1==值1,col_2==值2),"列名"]=value_2 # 注意是 df["col"].value df["列名"]=func(parameter_1,df["选择列名1"].value,df["选择列名2"].value,..)
def soc_iter(TEAM,home,away,ftr): df['Draws'] = 'No_Game' df.loc[((home == TEAM) & (ftr == 'D')) | ((away == TEAM) & (ftr == 'D')), 'Draws'] = 'Draw' df.loc[((home == TEAM) & (ftr != 'D')) | ((away == TEAM) & (ftr != 'D')), 'Draws'] = 'No_Draw' df["Draws"]=soc_loop("Arsenal",df["HOME"].value,df["AWAY"].value,df["FTR"].value)
2.2.9. 总结
总结: 如果必须要使用循环,建议使用 apply,同时考虑减少遍历元素数量和提前切片减小数据大小; 否则的话尽量适应向量化的方法
2.3. 高效数据拼接
2.3.1. 避免 append
数据在行层面的df.append,减少数据合并过程中的遍历搜索,建议使用先使用:
1. list.append 的方式添加到Dataframe中
2. 创建已知大小的空Dataframe,通过高效索引更改
# 第一种方式(运行时间最长——1分钟,内存占用一般) start1 = datetime.now() res1 = pd.DataFrame() for df in df_list: res1 = res1.append(df) print('append耗时:%s秒' % (datetime.now() - start1)) # %% 第二种方式(运行时间相对第一种少一些——46秒,但内存接近溢出) start2 = datetime.now() dict_list = [df.to_dict() for df in df_list] combine_dict = {} i = 0 for dic in dict_list: length = len(list(dic.values())[0]) for idx in range(length): combine_dict[i] = {k: dic[k][idx] for k in dic.keys()} i += 1 res2 = pd.DataFrame.from_dict(combine_dict, 'index') print('dict合并方式耗时:%s秒' % (datetime.now() - start2)) # 第三种方式:list装好所有值(运行时间最短——4秒多,内存占用低) start3 = datetime.now() columns = ['a', 'b'] a_list = [] b_list = [] for df in df_list: a_list.extend(df['a']) b_list.extend(df['b']) res3 = pd.DataFrame({'a': a_list, 'b': b_list}) print('list装好所有值方式耗时:%s秒' % (datetime.now() - start3))
2.3.2. join 代替 merge--x8加速
使用set_index 将需要merger的key column 列设置为dataframe 的index,然后使用df.join 合并确实可以加速。
加速前提
速度的提高将取决于您的索引是否唯一。如果索引不是唯一的,则在索引上合并两个数据帧可能甚至需要更长的时间
原理
Indices have a hash table. Meaning you can look them up in amortized O(1). For a normal column you need O(n) in worst case, meaning merging two dfs with len n takes O(n^2) in worst case
import pandas as pd import numpy as np myids=np.random.choice(np.arange(10000000), size=1000000, replace=False) df1 = pd.DataFrame(myids, columns=['A']) df1['B'] = np.random.randint(0,1000,(1000000)) df2 = pd.DataFrame(np.random.permutation(myids), columns=['A2']) df2['B2'] = np.random.randint(0,1000,(1000000)) %%timeit x = df1.merge(df2, how='left', left_on='A', right_on='A2') #1 loop, best of 3: 664 ms per loop %%timeit x = df1.set_index('A').join(df2.set_index('A2'), how='left') #1 loop, best of 3: 354 ms per loop %%time df1.set_index('A', inplace=True) df2.set_index('A2', inplace=True) #Wall time: 16 ms %%timeit x = df1.join(df2, how='left') ## df.join(other.set_index('key'), on='key') key A B 0 K0 A0 B0 1 K1 A1 B1 2 K2 A2 B2 3 K3 A3 NaN 4 K4 A4 NaN 5 K5 A5 NaN
2.4. 高效索引
pandas DataFrame 条件筛选是通过布尔索引完成的,其效率非常高。
2.4.1. 使用层次化索引筛选数据,避免使用条件逻辑筛选
层次化索引(hierarchical indexing)是pandas的一个重要的功能,它可以在一个轴上有多个(两个以上)的索引,这就表示着,它能够以低维度形式来表示高维度的数据。
df = DataFrame(np.arange(12).reshape(4,3, index=[["a","a","b","b"],[1,2,1,2]], columns=[["A","A","B"],["Z","X","C"]]) print(df) >>> ''' A B Z X C a 1 0 1 2 2 3 4 5 b 1 6 7 8 2 9 10 11 ''' #选取列,参数只能选取"A"或"B" print(data["A"])#等价于data.ix[:,"A"] ''' Z X a 1 0 1 2 3 4 b 1 6 7 2 9 10 ''' #选取行 print(data.ix["a"]) ''' A B Z X C 1 0 1 2 2 3 4 5 """
- df->Series,先把table_s转为层次化索引的Series,
- Series--索引-->数据。然后按索引值直接检索数据,
这比采用形如df[(df.col1==a)&(df.col2==b)]这种条件逻辑筛选法程序更简洁,运行也更快一些。
2.4.2. 正确使用 loc\iloc\ix\at
df.loc[i,c] 能够选取多行多列
df.at[i,c] 一次只能访问一个值,同等条件下比loc快10倍速
%%timeit outdf.loc[0] = indf.loc[0] >>> 100 loops, best of 3: 11.7 ms per loop %%timeit outdf.iloc[0] = indf.iloc[0] >>> 100 loops, best of 3: 11.4 ms per loop %%timeit outdf.ix[0] = indf.ix[0] >>> 100 loops, best of 3: 11.6 ms per loop %%timeit outdf.at[0,'time'] = indf.at[0,'time'] >>> 10000 loops, best of 3: 25.3 µs per loop
2.4.3. copy vs view
尽量使用切片而不是列表
df.iloc[:,10:20] # 更好 df.iloc[:,[10,11,...,20]] #差
2.5. 能改成numpy 就numpy
给numpy数据一个名字
我喜欢用 pandas, 因为 pandas 能让你给数据命名, 用名字来做 index. 在数据类型很多的时候, 名字总是比 index 好记太多了, 也好用太多了. 但是 pandas 的确比 numpy 慢. 好在我们还是有途径可以实现用名字来索引. 这就是 structured array. 下面 a/b 的结构是一样的, 只是一个是 numpy 一个是 pandas.
a = np.zeros(3, dtype=[('foo', np.int32), ('bar', np.float16)]) b = pd.DataFrame(np.zeros((3, 2), dtype=np.int32), columns=['foo', 'bar']) b['bar'] = b['bar'].astype(np.float16) """ # a array([(0, 0.), (0, 0.), (0, 0.)], dtype=[('foo', '<i4'), ('bar', '<f2')]) # b foo bar 0 0 0.0 1 0 0.0 2 0 0.0 """ def f1(a): for _ in range(N): a['bar'] *= a['foo'] def f2(b): for _ in range(N): b['bar'] *= b['foo'] print('%f' % ((t1-t0)/N)) # 0.000003 print('%f' % ((t2-t1)/N)) # 0.000508
2.6. 多线程并行计算
由于Pandas的一些操作都是单核的,往往浪费其他核的计算时间。
使用多CPU核心分块并行运算,对于可分块运行的数据运算任务,可使用多线程类编程,调用多个CPU核心并行工作提高速度。
2.6.1. multiprocessing.Pool
因为 GIL 的缘故 threading 不能用,那么我们就好好研究研究 multiprocessing。(当然,如果你说你不用 CPython,没有 GIL 的问题,那也是极佳的。)
首先介绍一个简单粗暴,非常实用的工具,就是 multiprocessing.Pool。如果你的任务能用ys = map(f, xs) 来解决,大家可能都知道,这样的形式天生就是最容易并行的,那么在 Python 里面并行计算这个任务真是再简单不过了。举个例子,把每个数都平方:
import multiprocessing import numpy as np import time def f(x): _sleep_time=np.random.rand()*10 time.sleep(_sleep_time) print ("sleep {} s".format(x,_sleep_time)) return x * x def f2(x): _sleep_time=x time.sleep(_sleep_time) print ("sleep {} s".format(x,_sleep_time)) return x * x cores = multiprocessing.cpu_count() pool = multiprocessing.Pool(processes=cores) xs = range(5) # method 0: built-in function map %%timeit print (list(map(f,xs))) >>> # 0 sleep 3.6515010268734596 s # 1 sleep 9.622811159423282 s # 2 sleep 4.5357524694481075 s # 3 sleep 0.06634438382474794 s # 4 sleep 9.26407616547759 s # [0, 1, 4, 9, 16] """ 有序输入,与形参无关的全局变量每次确定,返回有序 python-level loop """ print (list(map(f2,xs))) >>>... # 10s # method 1: map print (pool.map(f, xs)) >>> # 3 sleep 3.6515010268734596 s # 4 sleep 3.6515010268734596 s # 2 sleep 3.6515010268734596 s # 1 sleep 3.6515010268734596 s # 0 sleep 3.6515010268734596 s # [0, 1, 4, 9, 16] """无序输入,与形参无关的全局变量一次确定,返回有序 shuffle-map-reduce """ print (list(pool.map(f2,xs))) >>>... # 4s 多线程 分配计算,合并 # method 2: imap # 返回的是迭代器 print (list(pool.imap(f, xs))) # 0 sleep 4.854476661038866 s # 2 sleep 4.854476661038866 s # 4 sleep 4.854476661038866 s # 1 sleep 4.854476661038866 s # 3 sleep 4.854476661038866 s # [0, 1, 4, 9, 16] """无序输入,与形参无关的全局变量一次确定,返回有序 shuffle-map-reduce """ # method 3: imap_unordered # 返回的是迭代器 for y in pool.imap_unordered(f, xs): print(y) print (list(pool.imap_unordered(f, xs))) >>> # 2 sleep 2.1295588889839454 s # 1 sleep 2.1295588889839454 s # 0 sleep 2.1295588889839454 s # 3 sleep 8.694609405762515 s # 4 sleep 8.694609405762515 s # [0, 4, 1, 9, 16] """无序输入,与形参无关的全局变量分次确定,返回有序 shuffle-map """
当计算时间比较长的时候,我们可能想要加上一个进度条,这个时候 i 系列的好处就体现出来了。另外,有一个小技巧,就是输出 \r 可以使得光标回到行首而不换行,这样就可以制作简易的进度条了。
cnt = 0 for _ in pool.imap_unordered(f, xs): sys.stdout.write('done %d/%d\r' % (cnt, len(xs))) cnt += 1
3. 更小的pandas
# 查看内存占用情况 df.info() >>> <class 'pandas.core.frame.DataFrame'> Index: 1034849 entries, _14680064 to _6291451 Columns: 212 entries, 2016-01-01 to 2016-07-30 dtypes: float64(212) memory usage: 1.6+ GB
3.1. 降低数据精度
pandas中常用类型的数据类型内存占用大小情况:
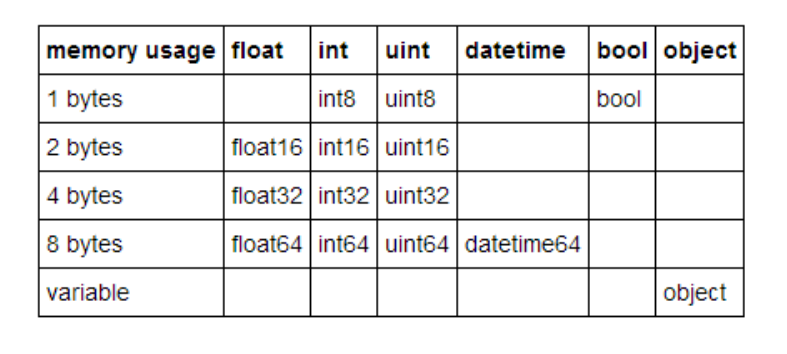
Pandas dtype mapping
| Pandas dtype | Python type | NumPy type | Usage |
| ------------ | ----------- | ---------- | ----- || | |
| object | str | string_, unicode_ | Text |
| int64 | int | int_, int8, int16, int32, int64, uint8, uint16, uint32, uint64 | Integer numbers |
| float64 | float | float_, float16, float32, float64 | Floating point numbers |
| bool | bool | bool_ | True/False values |
| datetime64 | NA | datetime64[ns] | Date and time values |
| timedelta[ns] | NA | NA | Differences between two datetimes |
| category | NA | NA | Finite list of text values |
主要思路
1. 将数值型列降级到更高效的类型(例如:float64->float32)
2. 将字符串列转换为类别类型
# float 类型 gl_float = gl.select_dtypes(include=['float']) converted_float = gl_float.apply(pd.to_numeric,downcast='float') #category (暂不推荐) #对于独特值少,重复值多的列有较好的效果 dow_cat = dow.astype('category')
3.1.1. 数据读取时
使用pd.read_csv()读取数据时,默认按64位精度读取数据,且有的数据读取为object类型,这些都极耗内存。可根据实际需要采用低位精度读取,并指定object类型为实际的类型或category类型,只读取需要处理的列。如
columns = ['O_LINENO','O_TERMINALNO','O_TIME','O_UP','O_NEXTSTATIONNO'] col_types = ['uint16','int32','category','int8','uint8'] columns_types = dict(zip(columns, col_types)) table_all = pd.read_csv(FOLDER + '\\' + 'table_all.csv',usecols=columns,dtype=columns_types) table_all.O_TIME=pd.to_datetime(table_all.O_TIME)
3.1.2. 优化数据结构
3.1.2.1. 稀疏矩阵
3.2. 优化字符串对象
3.2.1. 字符串内存占用组成
import sys # 空的二进制对象 val=b"" # 空的unicode对象 unicode_val=u"" sys.getsizeof(val) >>> 33 # 32+1 (nul) Python中的字符串是nul终止的 sys.getsizeof(unicode_val) >>> 49 # 48+1(nul)
pandas 中 :
# 内存占用情况 8 (PyObject*) + 48 (Python C struct) + string_length + 1
问题
pandas Dataframe 中可能存在(m,n)行列,数据巨大时候,意味着存在多个不长的字符串对象,每个字符串对象都 需要占用一个8 (PyObject*) + 48 (Python C struct)内存
df.shape >>> (1000000,1) # 10字符串长度 #10MB->./test.csv #60MB->df
4. 使用numpy 代替
我喜欢用 pandas, 因为 pandas 能让你给数据命名, 用名字来做 index. 在数据类型很多的时候, 名字总是比 index 好记太多了, 也好用太多了. 但是 pandas 的确比 numpy 慢. 好在我们还是有途径可以实现用名字来索引. 这就是 structured array. 下面 a/b 的结构是一样的, 只是一个是 numpy 一个是 pandas.
a = np.zeros(3, dtype=[('foo', np.int32), ('bar', np.float16)]) b = pd.DataFrame(np.zeros((3, 2), dtype=np.int32), columns=['foo', 'bar']) b['bar'] = b['bar'].astype(np.float16) # 5. a array([(0, 0.), (0, 0.), (0, 0.)], dtype=[('foo', '<i4'), ('bar', '<f2')]) # 6. b foo bar 0 0 0.0 1 0 0.0 2 0 0.0 def f1(a): for _ in range(N): a['bar'] *= a['foo'] def f2(b): for _ in range(N): b['bar'] *= b['foo'] print('%f' % ((t1-t0)/N)) # 0.000003 print('%f' % ((t2-t1)/N)) # 0.000508
可以看出来, numpy 明显比 pandas 快很多. 如果需要使用到不同数据形式, numpy 也是可以胜任的, 并且在还保持了快速的计算速度. 至于 pandas 为什么比 numpy 慢, 因为 pandas data 里面还有很多七七八八的数据, 记录着这个 data 的种种其他的特征。
5. 参考资料
-
How To Make Your Pandas Loop 71803 Times Faster https://towardsdatascience.com/how-to-make-your-pandas-loop-71-803-times-faster-805030df4f06 ↩
-
Stackoverflow:Performance of Pandas apply vs np.vectorize to create new column from existing columns ↩
-
Fast groupby Fast groupby-apply operations in Python with and without Pandas ↩
